mineXpert2
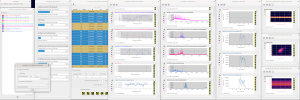
mineXpert2 est un logiciel libre sous license GPLv3+ qui facilite la visualisation et l’exploration de données de spectrometrie de masse MSⁿ.

mineXpert2 est un logiciel libre sous license GPLv3+ qui facilite la visualisation et l’exploration de données de spectrometrie de masse MSⁿ.
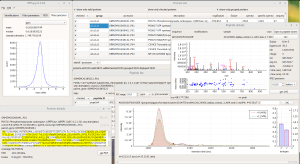
i2MassChroQ is a free software (GPL v3) that helps the user
i2MassChroQ is fully described in:
MCQR is an R package dedicated to statistical analysis of proteomic data. The goal of the MCQR is to provide a consistent set of functions facilitating proteomics data statistical analysis. It can be used without extensive programming knowledge with R.

MassChroQ (Mass Chromatogram Quantification) is a fast and versatile software that performs alignment, XIC extraction, peak detection and quantification on data obtained from Liquid Chromatography-Mass Spectrometry (LC-MS) techniques.
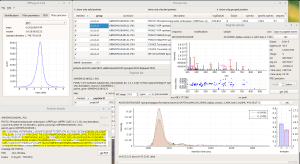
X!TandemPipeline has been renamed into i2MassChroQ